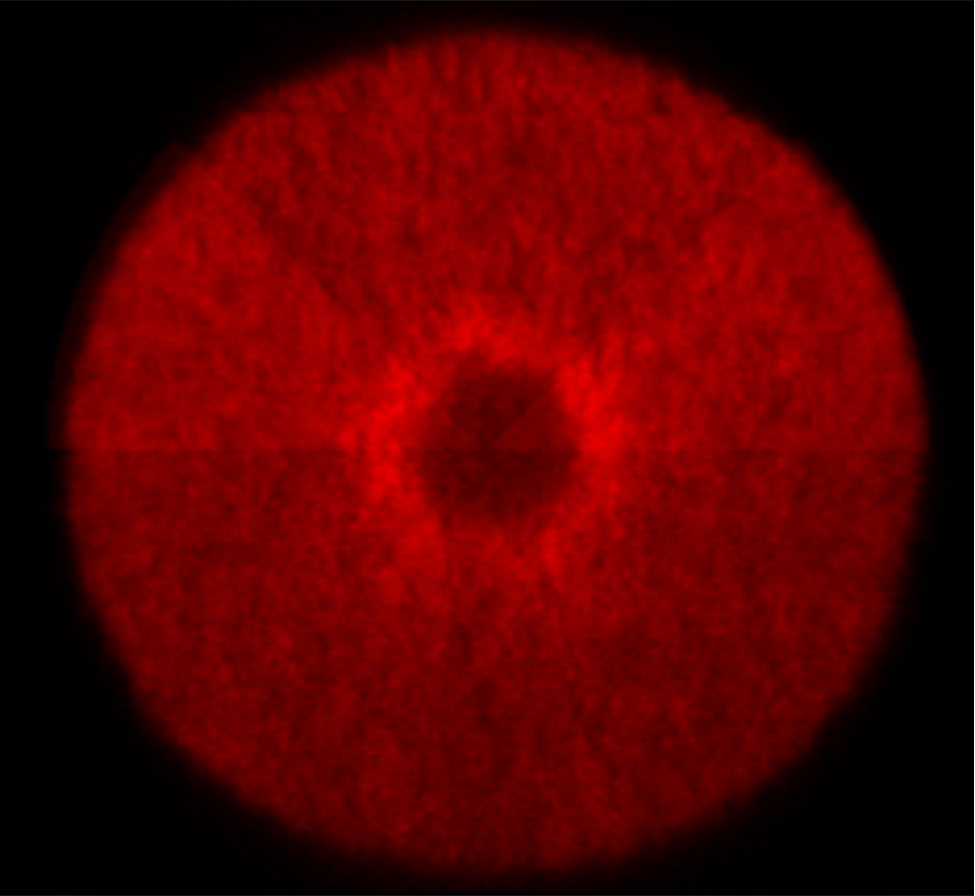
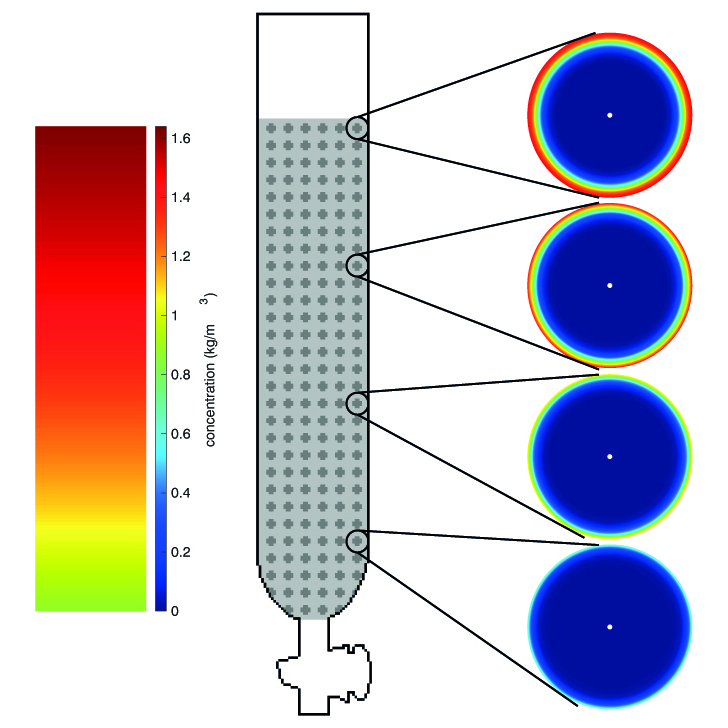
Im BMBF-Verbundprojekt AMSCHA entwickeln wir Modelle zur Simulation der Trennung von Protein- und Zellsuspensionen. Die Abkürzung des Projektes steht für »Analyse, Modellierung und Simulation von chromatographischen Aufreinigungsverfahren«. Gemeinsam mit Forschern der Rheinland-Pfälzischen Technischen Universität Kaiserslautern-Landau (RPTU) und der Hochschule Darmstadt werden auf verschiedenen Größenskalen Untersuchungen vorgenommen. Das Chemie- und Pharmaunternehmen Merck KGaA und der Hersteller von Lichtmikroskopen Leica Microsystems unterstützen das Forschungsprojekt seitens der Industrie.
Die Trennung von Zielstoffen aus einer Suspension ist ein wichtiger und oft unterschätzter Schritt bei der Wirkstoffherstellung in der pharmazeutischen Industrie. Dabei ist die Chromatographie als Trennverfahren sowohl im Labor als auch in der Industrie nicht mehr wegzudenken. Eine etablierte Form ist die Säulenchromatographie. Die Effizienz und der Durchsatz dieser Säulen hängt von den Prozessbedingungen und den verwendeten Chromatographiematerialien ab.